
Actas del Congreso Nacional de
Tecnología Aplicada a Ciencias
de la Salud



Actas del Congreso Nacional de Tecnología Aplicada a Ciencias de la Salud Vol. 2, 2019
En este trabajo se realiza la clasificación de tumores de mama mediante algoritmos K-means y GLCM para la extracción de características. Así como del algoritmo lineal support vector machine (SVM) para la clasificación de tumores en benignos y malignos en ecografías. Esto permitió clasificar correctamente al 84.7% de las ecografías.
Palabras clave: Ecografía ultrasónica, tumor, clasificación
In this work the tumor classification was done using K-means and GLCM algorithm to extract features. A lineal support vector machine (SVM) was used to classify in benign and malign tumors. 84% of echography were correctly classified
Keywords: Echography ultrasound, tumor, classification
La cuantificación de la información contenida en las imágenes biomédicas es uno de los aspectos de mayor interés, puesto que permite identificar estadios de normalidad y patológicos.
En el caso de la ecografía obtenida por ultrasonido este interés es mayor puesto que son pocos los trabajos que manipulan la imagen ecográfica para extraer información cuantitativa de la misma. En las ecografías las lesiones benignas tienden a ser más homogéneas y densas debido a la presencia de líquido. Los criterios médicos indican que los tumores benignos en su morfología tienden a ser ovalados, esféricos y elipsoidales mientras que las lesiones malignas tienden a ser más lobulares y espiculados, es decir suelen ser más heterogéneas debido a la alta probabilidad del desarrollo de calcificaciones.
Stavros et al [1] establecieron criterios para caracterizar masas mamarias sólidas; las que fueron clasificadas en tres categorías: benignas, indeterminadas y malignas. Para ser clasificado como tumor benigno un nódulo debe mostrar una de las tres características de benignidad: hiperecogenicidad homogénea; forma elipsoide, más ancha que alta, con orientación paralela a la piel; y dos o tres lobulaciones y con una cápsula ecogénica delgada. Por otra parte, un nódulo es clasificado como tumor maligno, cuando presenta características ecográficas como: contorno espiculado, más alto que ancho (orientación no paralela a la piel), márgenes angulados, marcadamente hipoecogénicos, con sombra acústica posterior, calcificaciones puntiformes, extensión ductal y microlobulación [2].
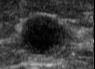
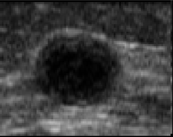
Analizamos la base de datos de ecografías de mama de Rodríguez Paulo Sergio [3], con el objetivo de clasificar las ecografías ultrasónicas de tejido mamario en benigno o maligno con el uso de algoritmos. Ejemplos de estas ecografías pueden verse en la figura 1, incisos a y c. Se calculó la matriz de concurrencia (GLCM) para pasar de 254 niveles de grises a 8 niveles, como puede observarse en la figura 1, incisos b y d. [4]
Analizamos la base de datos de ecografías de mama de Rodríguez Paulo Sergio [3], con el objetivo de clasificar las ecografías ultrasónicas de tejido mamario en benigno o maligno con el uso de algoritmos. Ejemplos de estas ecografías pueden verse en la figura 1, incisos a y c. Se calculó la matriz de concurrencia (GLCM) para pasar de 254 niveles de grises a 8 niveles, como puede observarse en la figura 1, incisos b y d. [4]
 a
a b
b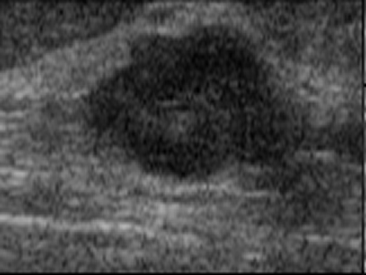 c
c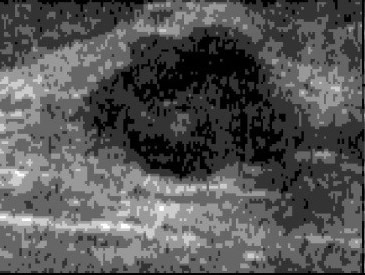 d
dFigura 1. (a) Ecografías original tumor benigno, (b) Matriz de co- ocurrencia tumor benigno de niveles de grises, .(a) Ecografías original tumor benigno, y (d) Matriz de co-ocurrencia tumor maligno de niveles de grises
Como puede observarse en la figura 2. Utilizando el algoritmo k-means se realizó clustering de los niveles de gris a 3 grupos como puede observarse en la figura 2.[2] Posteriormente se filtró la imagen para eliminar pequeños puntos y/o huecos; se estableció un tamaño de área de 1000 pixeles, para eliminar todas las área más pequeñas que esa cifra. Después de preprocesar las señales, se calcularon parámetros de interés como área, centroides, peso de los centroides, excentricidad, diámetro, numero de pixeles del área, eje mayor, eje menor, orientación y perímetro. Con estos datos se creó una de la base de datos.
 a
a b
b c
c d
dFigura 2. (a) Ecografía original, (b) clúster 1, (c) clúster 2 y (d) clúster 3
En la figura 3, incisos a y c se muestran las áreas que se detectaron como tumores y en los incisos b y d se muestran con color rojo algunos de los parámetros que se calcularon para dichas áreas.
 a
a b
b c
c d
dFigura 3. (a) área de tumor benigno. (b) parámetros calculados. (c) área del tumor maligno y (d) parámetros calculados
Se calcularon los parámetros de interés, pero sólo se exporta al archivo de Excel los correspondientes a la zona que se ha identificado como la correspondiente al tumor.
Se eligieron el parámetro eje mayor como variable de entrada y se realizó un análisis con el discriminante lineal y con las variables entradas, se clasificaron las imágenes.
De los 13 parámetros calculados se eligieron los 7 más representativos. Con el algoritmo SVM cubico, se clasificaron las 250 ecografías, con una efectividad del 84.7%. En la figura 4 se muestra la matriz de confusión, en la que se muestra el desempeño del algoritmo.
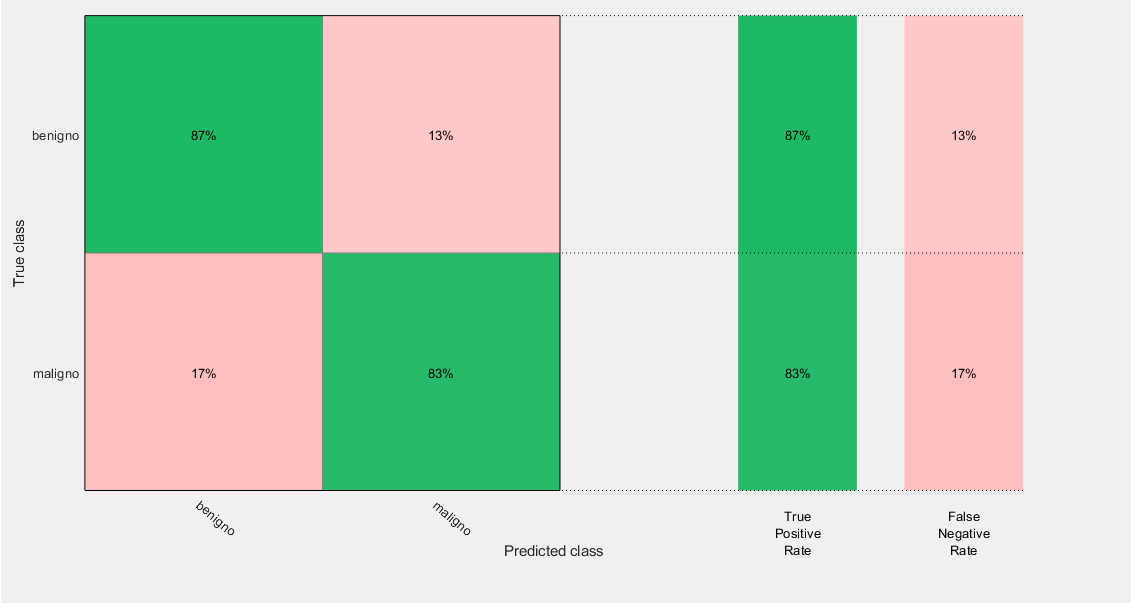
Figura 4. Matriz de confusión
La figura 5 muestra la clasificación utilizando en el eje X la variable del área y en el eje Y el diámetro. Con un círculo se indican las predicciones correctas y con una cruz las incorrectas. En azul los benignos y en color rojo los malignos.
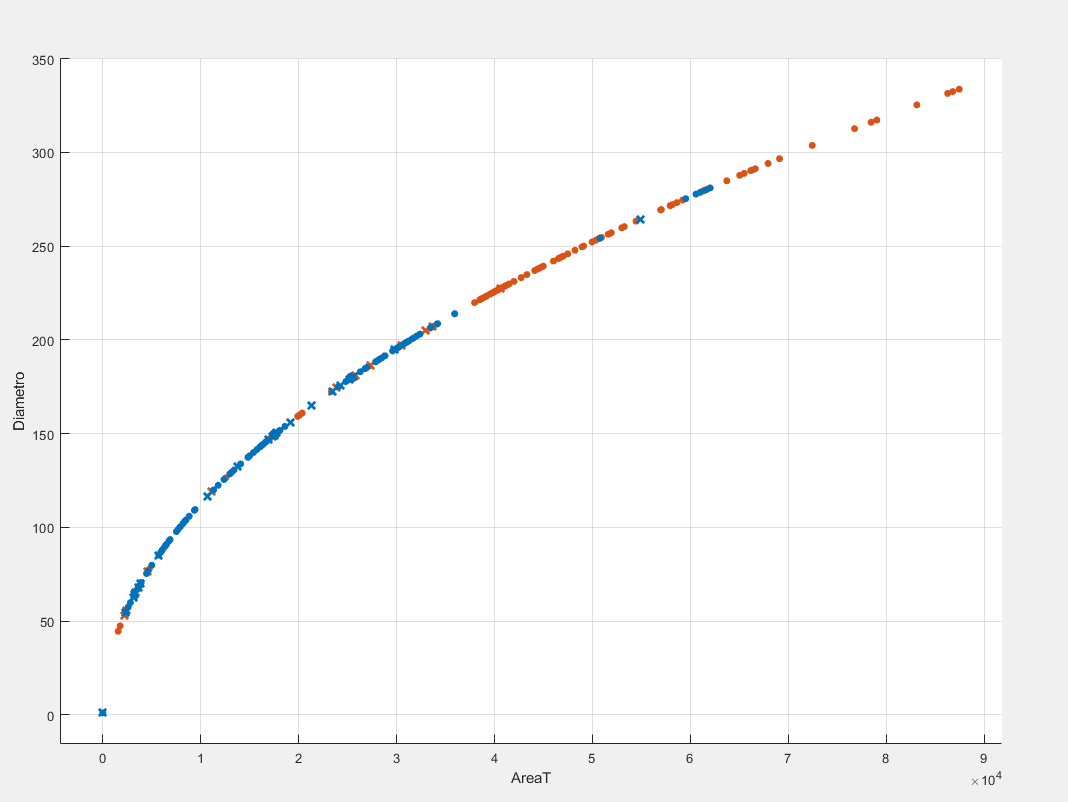
Figura 5. Clasificación de tumores en benignos y malignos
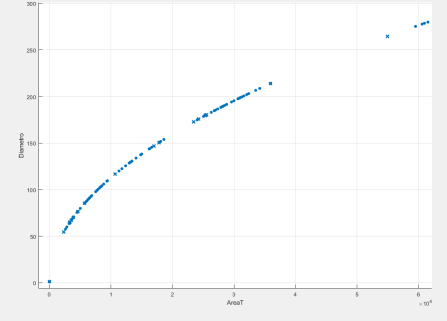
En las figuras 6 y 7, se muestra a detalle la clasificación correcta e incorrecta de los tumores.
 a
a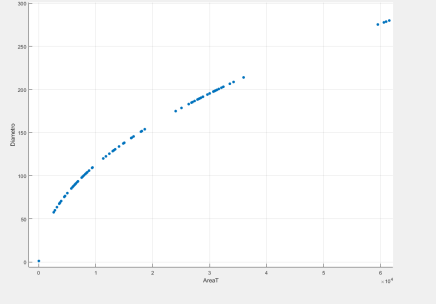 b
b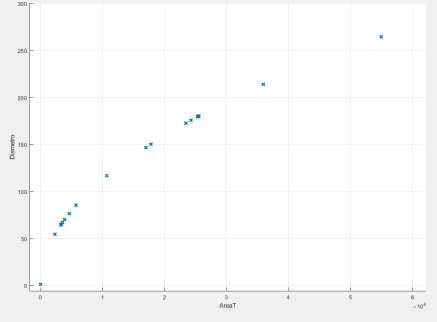 c
cFigura 6. (a) Clasificación de tumores benignos, (b) correctos y (c) incorrectos
 a
a b
b c
cFigura 7. (a) Clasificación de tumores malignos, (b) correctos y (c) incorrectos
El algoritmo de clasificación posee una capacidad de clasificación correctamente de clasificar correctamente del 84%.
En una imagen estándar de 8 bit, el número de niveles de gris es de 256, el ojo humano sólo puede detectar unas decenas de nivel de gris, por lo que los matices de gris resultantes de la distinta impedancia acústica de los tejidos pueden pasar desapercibidos. Por lo que clasificación automática de ecografías es un área de interés. De hecho, en la práctica clínica la interpretación de la imagen ecográfica es cualitativa y no cuantitativa, y el éxito depende de la pericia del técnico radiólogo y/o del médico y de su conocimiento de la normalidad.
La clasificación de ecografías presentadas en este trabajo es una alternativa para realizar un primer diagnóstico.
Los autores agradecen el apoyo de la DGAPA proyecto PAPIIT IG100419.