
Actas del Congreso Nacional de
Tecnología Aplicada a Ciencias
de la Salud



Actas del Congreso Nacional de Tecnología Aplicada a Ciencias de la Salud Vol. 4, 2022
En este trabajo aplicamos el método de convolución como procesamiento digital a imágenes de células HEK-293-ADENOVIRUS, desarrollando filtros por ventanas de 3X3 pixeles para la detección de bordes y contornos. Estos filtros de ventanas ya han sido usados para resaltar las regiones de interés en imágenes de muestras bilógicas para diagnóstico. Los filtros usados remarcan en algunos casos los bordes y contornos verticales u horizontales, se pueden aplicar mediante la convolución de matrices en imágenes en color y tonos de grises. En particular se aplicaron los filtros pasa bajos y altos, Mask, Frei&Chen y Sob&Kir con varios operadores en imágenes en tonos de grises.
Palabras Clave: Convolución de Matrices, Filtros, Imágenes Biológicas
In this work we apply the convolution method as digital processing to images of HEK-293-ADENOVIRUS cells, developing 3X3 pixel window filters for the detection of edges and contours. These window filters have already been used to highlight regions of interest in biological samples images to diagnostic. The filters used highlight in some cases the vertical or horizontal edges and contours, they can be applied by convolution of matrices in images in color and shades of gray. In particular, the filters passed low and high, mask, Frei&Chen and Sob&Kir were applied with several operators in shades of gray images.
Keywords: Matrix Convolution, Filters, Biological Images
En el proyecto de “Análisis de la expresión de interferón gamma humano usando el sistema de células HEK-293-ADENOVIRUS en ambientes de microgravedad”, auspiciado por el FOSEC, AEM-CONACYT del proyecto 262872, se desarrolló inicialmente para la instalación experimental de la instrumentación. Se construyó un sistema para capturar imágenes donde la resolución está restringida por el objetivo del microscopio, el campo de visión dado por el sensor de la cámara, la iluminación sobre las muestras, además del sistema de mecánico de enfoque y de la capacidad del sistema electrónico para el almacenamiento de las imágenes. El sistema se construyó como carga útil de un módulo aeroespacial para que la captura de las imágenes se diera en un ambiente de espacio cercano donde las propiedades fisicoquímicas no son tan propicias para su evolución o desarrollo de las células cancerígenas. El nombre “XIPE” proviene de la cultura mexica y representa en términos generales, la renovación, el desprendimiento de lo que ya no es útil, la regeneración de la naturaleza, así como del suelo seco en suelo fértil, conceptos que describen de manera general, algunos aspectos relacionados con la temática del proyecto que se describe en este trabajo y que será usado para describir al prototipo. El módulo experimental XIPE-1 es una plataforma tecnológica aeroespacial para la instalación y pruebas de experimentos científicos y desarrollos tecnológicos, Figura 1. No obstante, dada la versatilidad del instrumento, la funcionalidad del módulo permitirá dar cabida a otros experimentos científicos y tecnológicos que requieran validarse o evaluarse en condiciones de espacio cercano. Considerando principalmente bajas temperaturas (en algunos puntos cercanas a los 200 K bajo cero), baja presión barométrica, alta radiación solar y condiciones dinámicas de instalación mecánica (cercanas a los 2g).
En el proyecto de “Análisis de la expresión de interferón gamma humano usando el sistema de células HEK-293-ADENOVIRUS en ambientes de microgravedad”, auspiciado por el FOSEC, AEM-CONACYT del proyecto 262872, se desarrolló inicialmente para la instalación experimental de la instrumentación. Se construyó un sistema para capturar imágenes donde la resolución está restringida por el objetivo del microscopio, el campo de visión dado por el sensor de la cámara, la iluminación sobre las muestras, además del sistema de mecánico de enfoque y de la capacidad del sistema electrónico para el almacenamiento de las imágenes. El sistema se construyó como carga útil de un módulo aeroespacial para que la captura de las imágenes se diera en un ambiente de espacio cercano donde las propiedades fisicoquímicas no son tan propicias para su evolución o desarrollo de las células cancerígenas. El nombre “XIPE” proviene de la cultura mexica y representa en términos generales, la renovación, el desprendimiento de lo que ya no es útil, la regeneración de la naturaleza, así como del suelo seco en suelo fértil, conceptos que describen de manera general, algunos aspectos relacionados con la temática del proyecto que se describe en este trabajo y que será usado para describir al prototipo. El módulo experimental XIPE-1 es una plataforma tecnológica aeroespacial para la instalación y pruebas de experimentos científicos y desarrollos tecnológicos, Figura 1. No obstante, dada la versatilidad del instrumento, la funcionalidad del módulo permitirá dar cabida a otros experimentos científicos y tecnológicos que requieran validarse o evaluarse en condiciones de espacio cercano. Considerando principalmente bajas temperaturas (en algunos puntos cercanas a los 200 K bajo cero), baja presión barométrica, alta radiación solar y condiciones dinámicas de instalación mecánica (cercanas a los 2g).

Figura 1. Módulo del Microscopio XiPe-1
Por otro lado, el desarrollo tecnológico trae a veces consigo la extinción de algunos especialistas, como el caso de un cardiólogo que hacia sus diagnósticos con base a cardio-vectorgramas en 3D. En este sentido tenemos que ser claros que un sistema automático o computadora nunca reemplazará cien por ciento la interpretación o diagnóstico dado por un médico especialista, quien conoce el historial clínico y muchos demás detalles de sus pacientes. Entonces para el desarrollo de este trabajo tenemos presente que los médicos usan instrumentos para la auscultación superficial de sus pacientes, ejemplos de esto son termómetros, lámparas, estetoscopios, por mencionar algunos. Luego para analizar algunas enfermedades internas a veces se piden otros estudios invasivos y no invasivos, como análisis de sangre y en otros casos son imágenes biomédicas que dan soporte a su diagnóstico, [1,2]. Las imágenes capturadas tienen el problema de contraste y nitidez adecuados para hacer el análisis de población requerido para el estudio de diagnóstico y tratamiento del cáncer provocado por este tipo de células. Así en este trabajo se aplicó el método de convolución a las imágenes obtenidas del modelo de ingeniería, desarrollando filtros por ventanas o máscaras de 3X3 pixeles para la detección de bordes y contornos de las células. Los resultados obtenidos son las imágenes de las células cancerígenas en dos dimensiones con nitidez de bordes y contornos definidos para coadyuvar al médico experto en sus estudios para que puedan mejorar la ubicación, origen, interpretación, diagnóstico y tratamiento de enfermedades [3]. El procesamiento digital de imágenes permite, usando una imagen, obtener arreglos, suavizados, resaltes y otras modificaciones mediante técnicas de filtrado. Estas técnicas son filtros con los que se pueden obtener información detallada de una imagen como detección de bordes, contornos, [4].
Una de las operaciones más comunes sobre imágenes que se realiza en el dominio espacial es la convolución, en la cual un núcleo de números es multiplicado por cada uno de sus pixeles vecinos en una región pequeña, los resultados sumados obtenidos se colocan en sus lugares originales. Esto se aplica a todos los pixeles de la imagen, así en todos los casos los valores originales de los pixeles son multiplicados y sumados. Los valores obtenidos son usados para producir una nueva imagen, algunas veces estas operaciones se realizan en un lapso muy corto, donde las viejas imágenes son remplazadas por las nuevas. Para esto un núcleo de suavizado simple debe contener los valores como la Tabla 1.
Tabla 1. Máscara o núcleo para el operador del filtro de suavizado de 3x3
| 0.0625 | 0.125 | 0.0625 |
| 0.125 | 0.25 | 0.125 |
| 0.0625 | 0.125 | 0.0625 |
En realidad, esta implementación rápida debería ser para multiplicar los pixeles y sus 8 vecinos inmediatos por enteros 1, 2, o 4, la suma de productos, luego dividir el total entre 16. En este caso usando enteros que son potencias de 2 permite que el cálculo sea rápido (usando solo el bit de corrimiento), con un tamaño pequeño del núcleo (3x3) hace que la aplicación del algoritmo de suavizado sobre el dominio espacial de la imagen sea muy rápida [5].
Existen muchos núcleos en el dominio del espacio, incluyendo unos que toman las primeras derivadas (para detección de bordes y contornos) y las segundas derivadas (como el laplaciano, el cual es un operador no direccional que actúa como un filtro pasa altos para formar puntos y líneas). Estos son presentados generalmente como un conjunto de enteros con el entendido de que hay un divisor (usualmente igual a la suma de todos los valores positivos) que normaliza el resultado. Algunos de estos operadores pueden ser significativamente más grandes que el núcleo de 3x3 de la Tabla 1 mostrado anteriormente, introduciendo y sumando juntos la suma ponderada de los vecinos en una región más grande generalmente, pero no necesariamente cuadrada. Si se toma un núcleo más grande, lo cual implica que tomará más tiempo ya que se recorre como una plantilla sobre la imagen original. Aún si se tiene una computadora con gran poder de cálculo y un código no redundante (optimizado) para el proceso de multiplicaciones y sumas en un orden eficiente, el desempeño de la operación con un núcleo de 25x25 sobre una imagen de 512x512 pixeles se llevaría un tiempo considerable. La convolución en el dominio espacial es exactamente equivalente a la multiplicación en el dominio de frecuencia. Entonces una imagen es una función f(x,y) y el núcleo es g(x,y) describiremos la operación de convolución en la cual el núcleo está posicionado en cualquier parte de la imagen y multiplicado por ella como
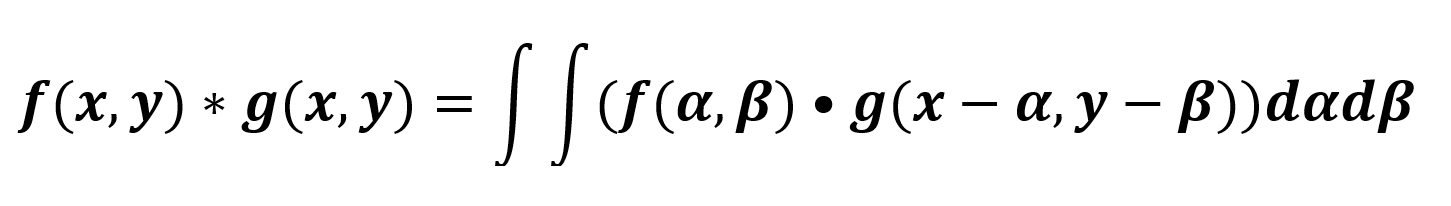
Donde α y β son variables pasivas para la integración, el rango el cual cruza la imagen entera, y el símbolo * indica la convolución. Si la transformada de Fourier de f(x, y) y g(x, y) son F(u,v) y G(u,v) respectivamente, entonces la operación convolución en el dominio de Fourier es simplemente la multiplicación de punto por punto, o
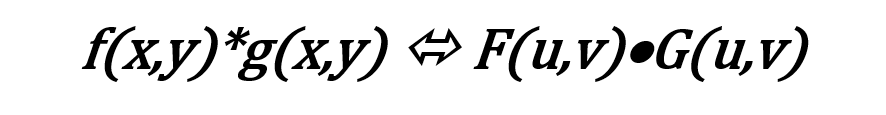
Existen unas cuantas diferencias practicas entre las dos operaciones, debido justamente a la aplicación de un núcleo en el dominio del espacio evita los pixeles de la orilla (estos son los más cercanos a la orilla de medio del núcleo), así sus vecinos no existen. Como una práctica alternativa, un núcleo diferente que es de un lado y tiene valores diferentes pesos puede ser aplicado a las orillas. La Figura 2 muestra ejemplos de convolución para suavizado de contornos.

Figura 2. Ejemplos de convolución para suavizado de contornos sobre imágenes digitales, primer renglón en color, segundo renglón en tonos de grises y tercer renglón suavizado a través de un filtro promedio
Puede notarse que la equivalencia de la multiplicación en el dominio de la frecuencia a la convolución en el dominio espacial está restringida a operadores con filtros lineales.
2.1 Convolución de matrices
En esta sección se explica breve y gráficamente en que consiste la convolución de las matrices que se emplean para hacer el procesamiento de las imágenes de muestras biológicas. Así entonces una imagen digital puede representarse como la expresión z = f(x,y) en 2-D, donde x: abscisas e y: ordenadas, son las coordenadas espaciales y z es el parámetro que proporciona el valor de la intensidad de un punto (x,y) en la imagen. Por otro lado, las imágenes analógicas son siempre funciones continuas. Cuando se requiere digitalizar una imagen se lleva a cabo un proceso de discretización tanto para las coordenadas como para la intensidad, similar a como se hace en una señal analógica. En procesamiento de señales la digitalización de las coordenadas se denomina muestreo, lo mismo en imágenes y a su vez la digitalización de la intensidad se denomina cuantización. Esto puede ser útil en imágenes biológicas para soporte de diagnóstico, aplicando métodos que implican modificar píxel por píxel para resaltar bordes y contornos mediante la aplicación de filtros [6].
En el caso de una imagen digital en escala de grises el modelo representativo de la matriz está dado por zij = f(xi,yj). En donde las entradas de la matriz son pixeles y el número total de pixeles es el orden de la matriz, a mayor número de pixeles mejor calidad de la imagen, pero también mayor el tiempo para procesarla y sabemos también que el orden define la resolución de la imagen digital. Luego los valores en cada pixel están cuantificados por un número de bits desde 0 a 2p-1 posibles dependiendo del formato de la imagen que proporciona la calidad de imagen, blanco y negro, tonos de grises o los planos RGB. Generalmente en imágenes se usa un p=8, por lo que el intervalo de intensidades varía entre 0 y 255. En particular si se emplea una imagen digital coloreada se subdividen en los tres planos R, G y B que corresponden a los colores rojo, verde y azul con sus respectivos valores de intensidades. Cuando se aplican estas máscaras a las matrices de las imágenes mediante operadores, al procedimiento se le llama convolución de matrices. Primero entiéndase a estas matrices como arreglos de mxn que cumplen con las mismas propiedades de las matrices. Esto puede representarse matemáticamente como sigue: dado un arreglo Amxn y una máscara C(2N+1)x(2N+1) con 2N + 1 < m, n; se denomina la convolución de los arreglos A y C como un nuevo arreglo D = A*C que puede escribirse a partir de la ecuación
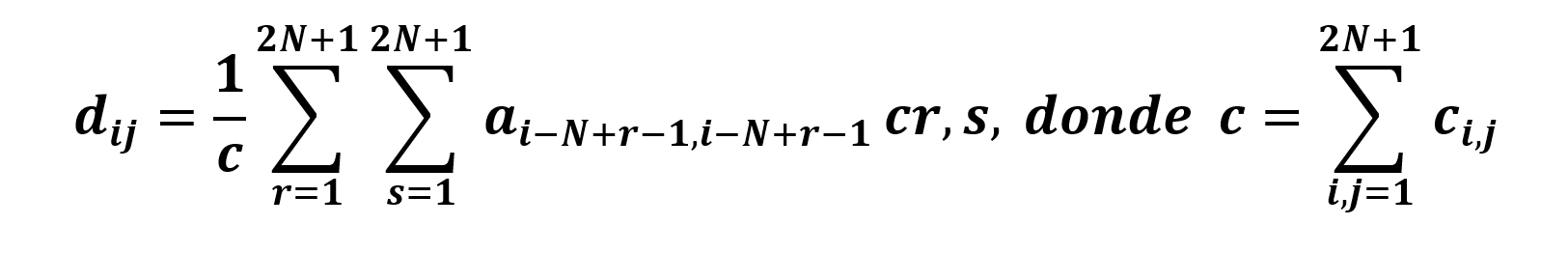
(tomando a c=1 si c=,0 para evitar la indeterminación). Obsérvese que dij está definido para i=N+1,…,m-N-1 y j=N+1,…,n-N-1. Por lo tanto, al arreglo o matriz C se le conoce como el núcleo de la convolución. En las dos siguientes Figuras se muestra el proceso de convolución de una matriz dada por otra de orden 3x3. La Figura 3 recoge el resultado de la convolución correspondientes a las entradas (2, 2), (2, 3) hasta la (4, 4) y también se muestra el resultado final de la convolución, con las entradas de la primera-última fila y primera-última columna manteniendo los valores originales.
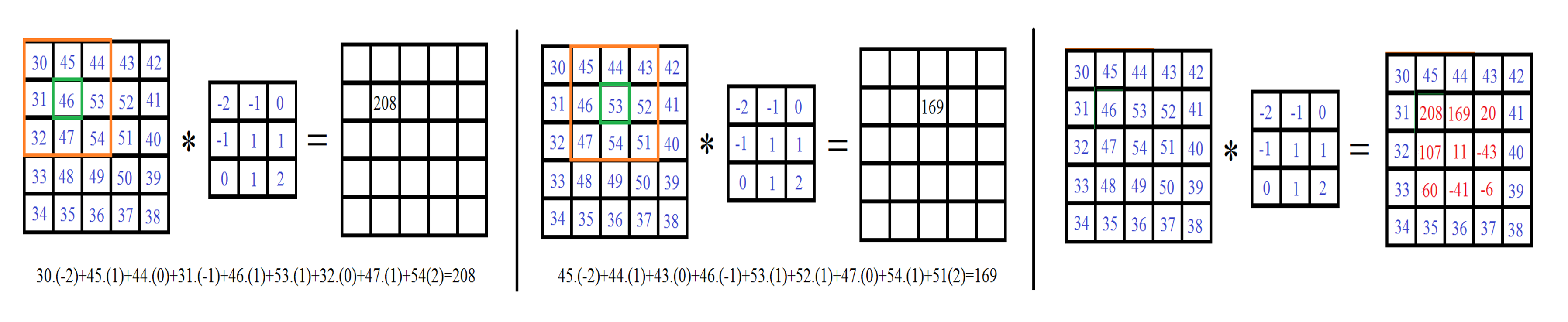
Figura 3. Proceso de la Convolución mediante máscaras de matrices
2.2 Similaridad y Correlación Normalizada
La similaridad entre dos imágenes es una cantidad mesurable mediante la correlación de normalización cruzada definida como:
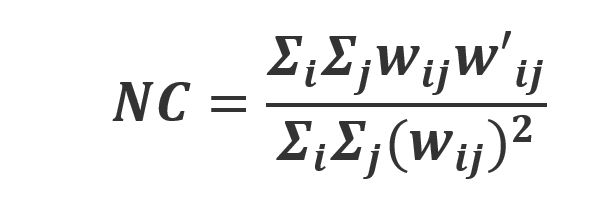
donde Wij y W'ij representan los valores de cada píxel en la posición (i, j), en las imágenes biológicas original y en la imagen filtrada, Figura 2. Este procedimiento muestra que tanto alteración y modificación se presentan en la imagen después de aplicar la convolución mediante operadores de máscara, [7,8]. En algunos casos las imágenes biológicas puras, es decir sistemas que no usan filtros, se ven involucrados en algún tipo de problema para la aplicación de operadores máscara para la convolución. Un caso de esta problemática es la similitud, es decir, que pasa si las máscaras tienen cierta similitud con las imágenes originales. Por tal razón la similitud se define como sigue: sean S={C,M,E,D}, donde C es la imagen original, M la máscara, E la imagen filtrada y D el método de convolución. El problema se presenta justamente cuando C y E son perceptualmente similares [9,10], en este sentido se define la similitud como. - sea C un conjunto no vació, entonces una función sim: C2→(-∞,1] se denomina función C si para x e y ∈ C.
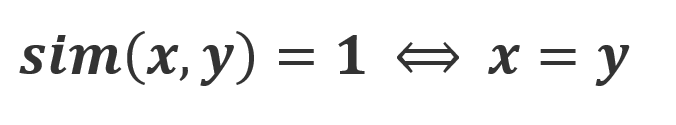
Para x≠y, si sim(x, y) <1, un ejemplo de esto sería:
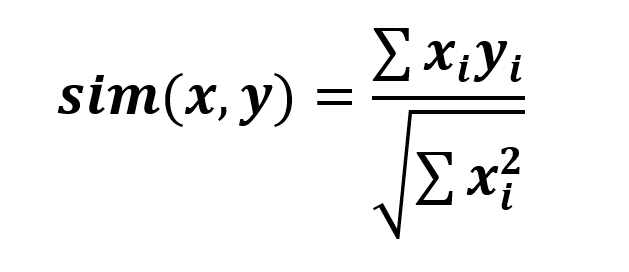
Condición práctica: sim(C,E(c, m))≈1 para toda m∈M y c∈C. Para la selección de la imagen se debe seleccionar una imagen que no se modifique significativamente como resultado del proceso de convolución.
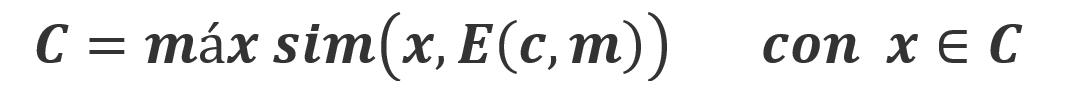
Una vez que se desarrollan los diferentes operadores de convolución para aplicar las técnicas de filtrado mediante matrices (también llamados núcleos o máscaras) en arreglos de 3x3 pixeles, nos proponemos a aplicarlos a imágenes digitales biológicas de 512x512 pixeles, a 300 pixeles por pulgada que obtuvimos del microscopio XiPe-1. Entre las cuales están una imagen del cultivo de las células HEK-93 a una distancia focal 10x y otra a 20x. Se toma cada imagen digital y se convierte en tonos de grises con el objeto de trabajar en un solo plano, se hace recorrer una máscara 3x3 pixeles de cada operador para hacer las operaciones requeridas en el dominio del espacio para la convolución y obtener el filtrado de la imagen. Los operadores desarrollados se encuentran en la Figura 4 y en la Tabla 2 se contabilizan.

Figura 4. Matrices núcleo para los operadores de convolución
Tabla 2. Operadores de filtrado para la convolución
| Operador | No. Filtros | Operador | No. Filtros | Operador | No. Filtros |
| Smooth | 1 | Sobel | 2H y 2V | Passbass&high | 3 pb y 3 ph |
| Media | 1 | Derivada | 3 | Mask | 7 |
| Prewitt | 2H y 2V | Sharpen | 3 | Frei&Chen | 9, 1H y 1V |
| Roberts | 2H y 2V | Laplace | 4 | Sob&Kir | 12 |
Cuando se obtienen las imágenes de convolución usando los operadores de filtrado y las máscaras de la Tabla 2, se procede a generar las imágenes de la diferencia entre las de convolución y las originales. Se mostrarán las gráficas obtenidas para el número de filtros contra la Normalización Cruzada y contra la Similaridad de la diferencia de imágenes de los filtros para algunos operadores. Luego se dan las interpretaciones de las gráficas obtenidas tomando un valor de umbral como referencia. Se desarrolla una breve discusión entre toma de imágenes invasivas y no invasivas, luego se anotan las conclusiones del trabajo en el sentido de la aportación en el área del comportamiento de los cultivos en condiciones extremas.
3.1 Resultados
Se aplicaron todos los operadores de filtrado y cada una de las máscaras, un total de 54, a las 2 imágenes biológicas, es decir 108 imágenes de convolución y otras tantas para la diferencia con las originales. Sin embargo, siguiendo el procedimiento se muestran solo las imágenes digitales que en nuestro caso obtuvieron mejor calidad y nitidez, resaltando los bordes y contornos para las máscaras empleadas, Figura 5. Los operadores con mejores resultados de convolución y diferencia se muestran en la Tabla 3.
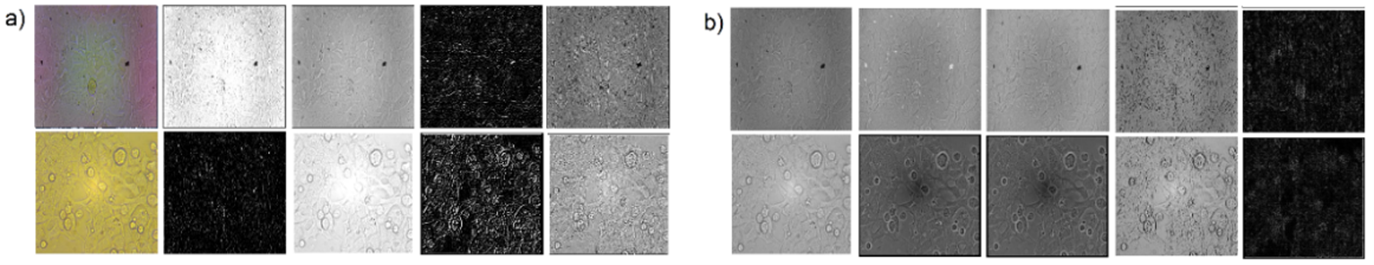
Figura 5. a) Algunas imágenes de convolución, b) diferencias entre la original y la filtrada, 1er fila: 10x, 2a fila: 20x
Tabla 3. Mejores resultados obtenidos por los operadores y las diferencias empleados en las células HEK-293
| Operadores de Convolución | Células 10x | Células 20x |
| Sobel | Sob 1, dif 2 | Sob 1, dif 2 |
| Sharpen | Shar 2, dif 2 | Shar 2, dif 2 |
| Laplace | dif_1 | Lap 3, dif 1 |
| Passbass&high | pb_4, dif 6 | Pb 5, dif 6 |
| Mask | dif 5 | dif 6 |
| Frei&Chen | Frei 10, dif 1 | Frei 11, dif 1 |
| Sob&Kir | S&k 12, dif 6 | S&k 8, dif 8 |
Ahora mostraremos las gráficas para cuatro operadores de filtrado de la Normalización Cruzada y la Similaridad entre las imágenes de convolución y las imágenes de las diferencias, Figuras 6 y 7 respectivamente.
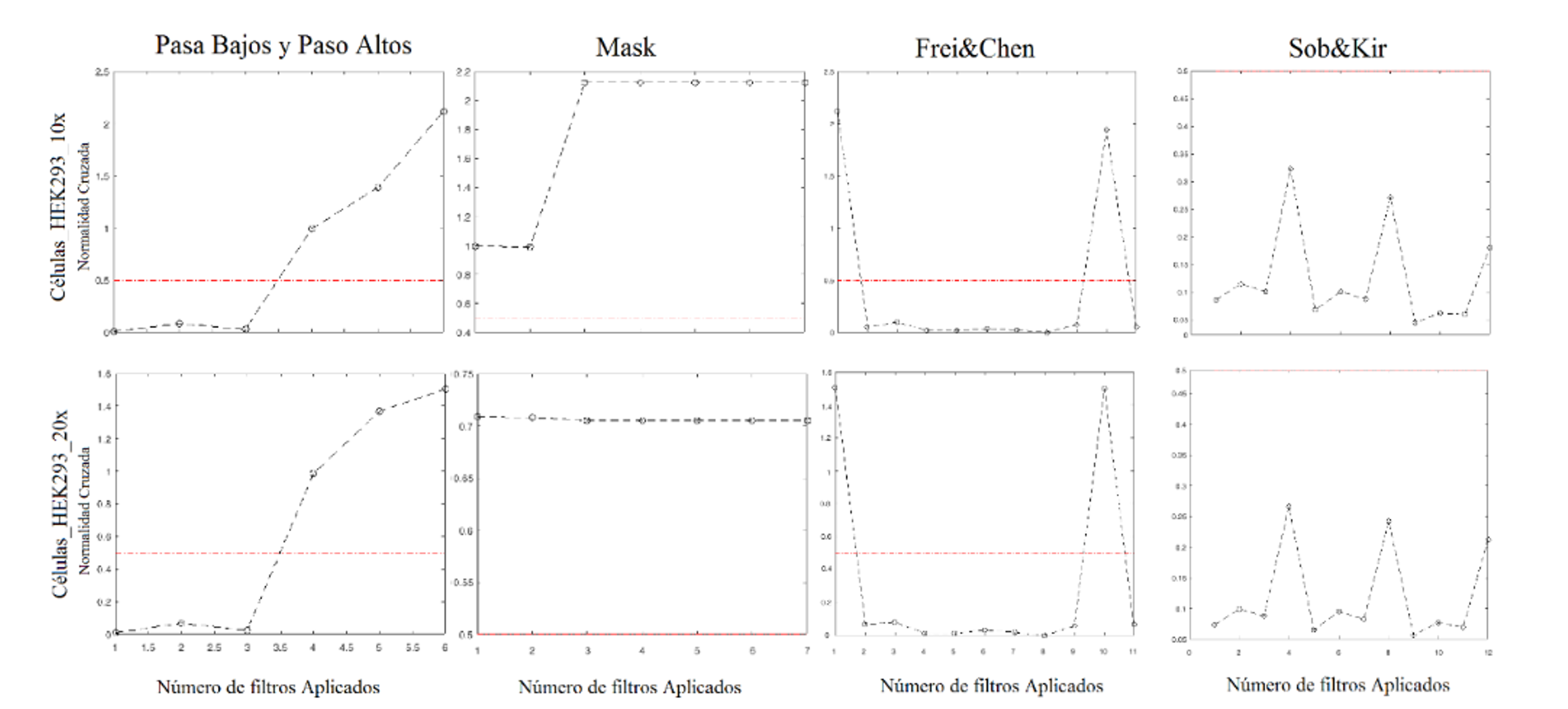
Figura 6. Normalidad Cruzada de los operadores Pasa bajos y altos, Mask, Frei&Chen y Sob&Kir para el angiograma del ojo y la mastografía
En la Figura 6 se muestra como en la Normalidad Cruzada hay valores por debajo del umbral para los filtros Pasa Altos, Frei&Chen y Sob&Kir. En el caso de valores por arriba del umbral están en los operadores Pasa Bajos, Mask pocos en Sob&Kir. En la Figura 7 se muestra como en la Similaridad entre imágenes de convolución y las originales casi todos los valores están por debajo del umbral=0.5 lo que indica que si hay gran diferencia entre imágenes originales y filtradas.
Si bien las técnicas de filtrado son clásicas y la mayoría de los casos ya están implementadas en los sistemas de adquisición de imágenes, sólo los filtros estándares, en este trabajo aplicamos el método de convolución mediante las máscaras para los operadores de filtrado con los cuales se obtuvo mejores resultados en resaltar bordes y contornos. Sin embargo, se mostró también que en algunos casos las diferencias entre las imágenes originales y las filtradas tienen mejor contraste y nitidez que nos sirven para resaltar mejor las células cancerígenas. Esto como lo mencionamos desde un principio puede dar soporte a una prescripción o conteo de los cultivos de este tipo de células para dar un diagnóstico o tratamiento. Se presentaron gráficas de la normalidad cruzada y similaridad bajo un umbral para tener una medida de la semejanza entre una imagen original y una filtrada.
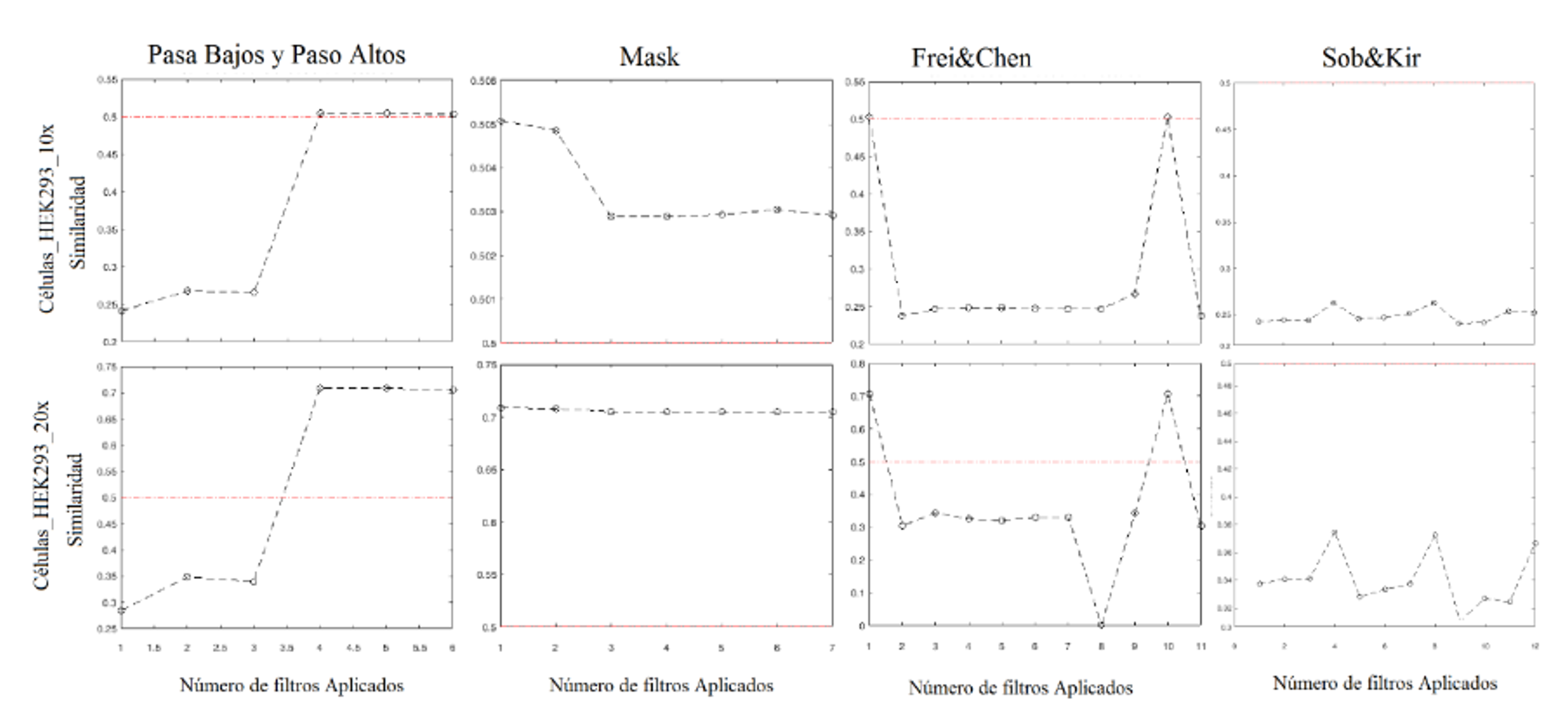
Figura 7. Similaridad de los operadores Pasa bajos y altos, Mask, Frei&Chen y Sob&Kir para el angiograma del ojo y la mastografía